大家好,欢迎来到IT知识分享网。
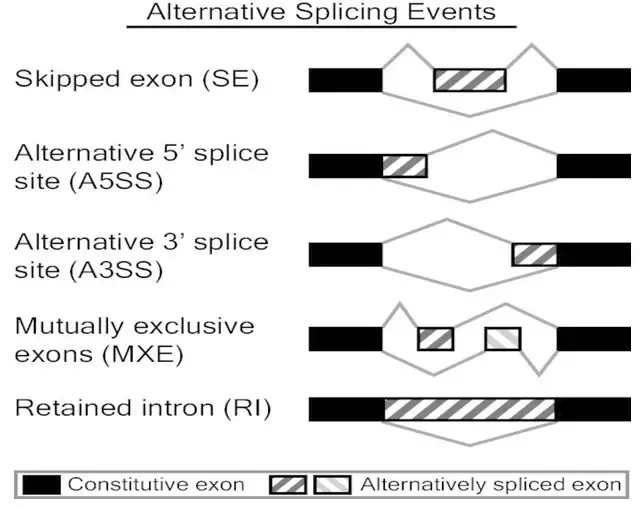
可变剪切的概念
可变剪切是指从一个mRNA前体中通过不同剪接方式,选择不同的剪接位点组合,所产生不同的mRNA剪接异构体的过程。
可变剪切的分类:
- 外显子缺失 (Exon skipping);
- 可变的5’端剪切 (Alternative 5’ splicing);
- 可变的3’端剪切 (Alternative 3’ splicing);
- 互斥外显子 (Mutually exclusive exons);
- 内含子包含 (Intron retention)。
可变剪切模型预测
应用SpliceGrapher软件,可以根据转录组测序得到的序列和参考基因组的注释文件生成可变剪切模型。
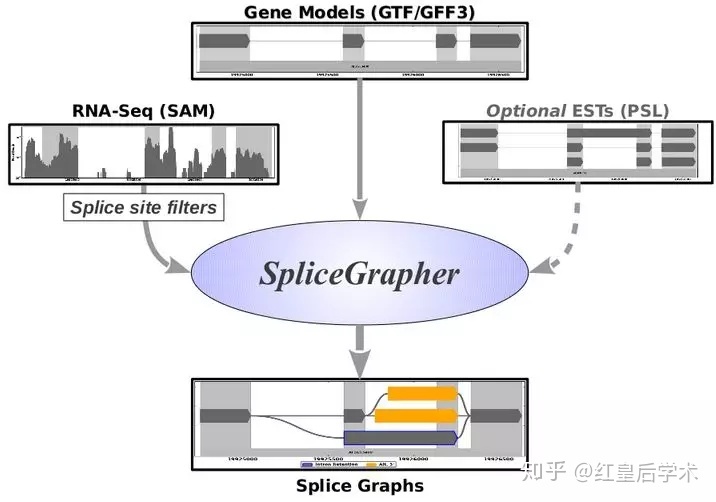
该分析需要两种输入文件,参考基因组注释gtf/gff3文件和测序reads与参考基因组比对的sam文件。
SpliceGrapher软件的分析过程比较简单,共分包括4个步骤:
首先,应用SpliceGrapher包含的物种特性筛选文件,去除sam文件中的假阳性可变剪切;
然后用筛选后的sam文件预测可变剪切模型;
第三步需要用参考基因组的注释gtf/gff3文件产生已知的可变剪切模型;
最后,用SpliceGrapher的绘图脚本将可变剪切模型进行可视化。
结果解读
一个张图片为一个基因的模型,左下角和右下角的数字分别代表该基因的起始位点和终止位点。
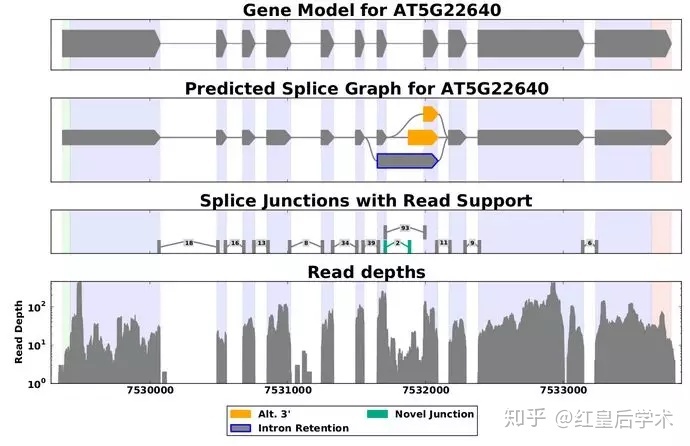
图中共包括4行,第一行是该基因的注释文件中所包含基因模型,第二行为根据测序结果与注释文件共同预测出的基因模型,第三行内容与第二行一致,不同的是只画出具有代表性的转录本,第四行则是测序文件中匹配到各个区域的reads数目。
图中五边形代表外显子,它们之间的连线表示不同的剪接方式。
紫色背景表示有外显子出现的区域,白色背景表示内含子区域。
图中白色背景的宽度并不代表真实的内含子长度,有些基因内含子区域远比外显子区域长,为了更清楚地展示可变剪切模型,内含子区域会被缩短很多。
不同的可变剪切事件用不同的颜色标注出来,并在图片下方的图例中给予解释。

免责声明:本站所有文章内容,图片,视频等均是来源于用户投稿和互联网及文摘转载整编而成,不代表本站观点,不承担相关法律责任。其著作权各归其原作者或其出版社所有。如发现本站有涉嫌抄袭侵权/违法违规的内容,侵犯到您的权益,请在线联系站长,一经查实,本站将立刻删除。 本文来自网络,若有侵权,请联系删除,如若转载,请注明出处:https://haidsoft.com/139570.html

