大家好,欢迎来到IT知识分享网。
文章信息

摘要:尽管生物多样性丧失问题普遍存在,但我们对于物种和种群将如何应对加速的气候变化仍然知之甚少。在本研究中,我们整合了种群基因组学、实验进化和环境建模等方法,以阐明大叶杨(Populus lasiocarpa)对气候变化的进化响应。大叶杨是一种关键的高山森林树种,主要分布在一个全球生物多样性热点地区的山区。研究发现,在历史时间尺度上,种群动态、分化选择和长期平衡选择共同塑造并维持了种群内部和种群之间的遗传变异。在研究当代气候适应的基因组特征时,我们发现单倍型块(可能由抑制重组的倒位多态性引起)与富集的局部适应性环境变异组合相关。我们进一步评估了环境诱导的可塑性响应、遗传集群间的组成型表达差异及其相互作用在驱动基因表达变异和分化中的相对贡献。值得注意的是,我们观察到遗传集群间的序列分化与组成型差异表达之间存在强相关性。最后,通过将遗传适应、迁移和遗传负荷纳入种群水平的气候变化风险预测中,我们确定西部种群 —— 主要分布在横断山区(该地区以环境异质性和显著的生物多样性著称)—— 是最易受气候变化影响的种群。这些种群应作为保护和管理的优先对象。总体而言,本研究增进了我们对长期自然选择、局部环境适应和即时可塑性表达变化在塑造关键物种自然种群应对气候变化响应中相对作用的理解。
引言
人为气候变化及相关的环境改变已导致物种分布范围迁移、种群数量下降乃至灭绝,成为全球生物多样性丧失的一个重要驱动因素。在此背景下,了解并监测当地生态系统中的关键物种如何应对快速气候变化,是生物多样性保护的关键优先事项(Urban 等,2016)。基因组数据是记录过去环境波动中遗传适应情况的主要载体之一,也为预测气候变化背景下未来适应的进化潜力提供了一种潜在的有用工具(Bernatchez 等,2024;Feng 等,2024)。我们需要整合生态学和气候基因组学,以更好地理解和预测气候变化对基因组、种群和物种水平多样性的影响(Martin 等,2023)。
面对前所未有的环境变化,自然种群可以通过基于遗传的进化改变在原地存续,迁移到更适宜的地点(对于植物而言,这取决于花粉和种子的传播能力),和 / 或依靠环境诱导的表型及基因表达可塑性(Alberto 等,2013)。反之,它们可能面临灭绝的风险。在遗传适应性进化方面,包括突变、自然选择和重组在内的各种进化过程,在塑造对气候变化的响应中发挥着关键作用(Aguirre-Liguori 等,2021)。快速应对环境变化的能力往往可能取决于现有的适应性遗传变异量,包括适应性单核苷酸多态性(SNPs)、短插入 / 缺失(indels)和大型结构变异(SVs)。这些变异在很大程度上由进化因素维持,如突变、遗传漂变、基因流以及选择压力,包括种群内部和种群之间的长期平衡选择和 / 或分化选择(DS)(Olson-Manning 等,2012)。值得注意的是,一种结构变异 —— 倒位多态性,已被反复证实与物种的局部生态适应相关,例如拟态蝴蝶(Joron 等,2011)、猴面花(Lowry 和 Willis,2010)、向日葵(Todesco 等,2020)和鹿鼠(Harringmeyer 和 Hoekstra,2022)。倒位会抑制杂合子中的重组,有助于在不利基因流的情况下,将一组局部适应的等位基因保持在一起(Faria 等,2019)。
此外,基因表达变异对环境影响高度敏感,因此,环境诱导的表达可塑性被认为对促进快速环境变化下的即时响应至关重要(Josephs 等,2021;Swaegers 和 Koch,2022;Coates 等,2025)。随着种群适应动态且多样的环境,自然选择可能会推动自然种群间适应性组成型表达差异的出现和确立(Carruthers 等,2022;Bhaskara 等,2023)。越来越多的证据表明,表达差异并非完全是可塑性的,而是部分受遗传控制(Jacobs 等,2020;Wos 等,2021;Szukala 等,2023;Durkin 等,2024)。然而,序列进化与表达差异之间的关系仍知之甚少(Ballinger 等,2023;Bhaskara 等,2023)。了解组成型和可塑性表达差异的相对贡献及其相互作用,对于阐明自然种群在适应快速环境变化过程中基因调控的动态至关重要(Ghalambor 等,2015;Campbell-Staton 等,2021;Wood 等,2023)。填补这一知识空白,对于确定基因表达的进化如何驱动局部适应并塑造自然种群对气候变化的响应也至关重要(Sandoval-Castillo 等,2020;Oomen 和 Hutchings,2022)。
森林生态系统为生物多样性提供栖息地,并为人类提供环保产品。树木作为生态系统的构建者,通过高效发挥碳汇功能,在全球碳循环中发挥着关键作用(Isabel 等,2020)。然而,由于树木寿命长,大多数树木在跟上快速气候变化方面面临重大挑战,尤其是考虑到这种威胁可能在单个个体的生命周期内显现(Holliday 等,2017;Browne 等,2019)。东南亚山区环境高度复杂且异质,被公认为全球生物多样性热点地区,以极高的物种特有性著称(Myers 等,2000)。但环境波动加剧,使得该地区的物种在未来气候变化情景下特别容易出现适应不良(Chen 等,2022)。大叶杨(Populus lasiocarpa)是一种优势且特有的雌雄异株树种,因其异常大的叶片而在杨属中脱颖而出。该物种主要分布在东南亚四川盆地周边的山区(Li 等,2023)。大叶杨的自然种群见于横断山区(西部)、秦岭(北部)、大巴山和巫山(东部)以及云贵高原(南部)(图 1a)。这种环状地理分布使大叶杨及其自然种群成为研究种群历史、地理和气候变化如何相互作用以塑造物种内种群分化的绝佳对象(Davis 和 Shaw,2001;Davis 等,2005)。鉴于这一关键物种的生态重要性,迫切需要全面预测和评估种群层面对未来气候变化的脆弱性,特别是该生物多样性热点地区的特有林木。
在本研究中,我们构建了大叶杨的从头染色体水平参考基因组,随后获取了来自其天然分布范围内 22 个种群的 200 个个体的全基因组重测序数据(图 1a;补充图 S1,在线补充材料)。此外,我们还收集了来自代表性西部和东部种群的实验幼苗在对照和胁迫条件下叶片和根组织的 RNA-seq 数据,这些种群分别栖息在不同的热力和水涝环境中。利用这一全面的基因组和转录组数据集,我们的目标如下:(i)研究种群历史、地理和自然选择如何影响基因组变异模式;(ii)剖析促进局部环境适应的遗传基础并识别潜在的适应性变异;(iii)探究环境诱导的可塑性以及基因表达变异的组成型分化成分的程度,以及它们与遗传变异和序列进化的关系;(iv)预测种群未来对气候变化的脆弱性,并确定那些风险最高且需要紧急管理措施的种群。
结果
大叶杨高质量参考基因组组装
本研究采用多方面测序方法,包括 49.95 Gb 的纳米孔测序读长(约 119×)、36.37 Gb 的 Illumina 读长(约 87×)以及 62.52 Gb 的高通量染色体构象捕获(Hi-C)读长(约 149×),用于大叶杨染色体级别的基因组组装(补充表 S1,在线补充材料)。经过初始基因组组装、多轮 polishing 及冗余序列过滤(补充表 S2,在线补充材料)后,最终组装获得了 419.54 Mb 的基因组序列,contig N50 大小为 9.19 Mb,99.23% 的组装序列被锚定到 19 条假染色体上(补充图 S2 及补充表 S3,在线补充材料)。通过对 BUSCO(Benchmarking Universal Single-Copy Orthologs,Simão 等,2015)单拷贝直系同源基因的召回率(分别为 97.9% 和 97.8%)以及 Illumina 短读长比对率的验证,证实了组装基因组的完整性和准确性(补充表 S4 和 S5,在线补充材料)。
随后,我们对重复元件、蛋白质编码基因和非编码 RNA 进行了注释。转座元件(TEs)共占基因组的 40.2%,其中反转录转座子和 DNA 转座子分别占基因组的 20.8% 和 15.9%(补充表 S6,在线补充材料)。通过结合同源预测、转录组预测和从头预测的综合策略,共注释得到 39,008 个高置信度的蛋白质编码基因(补充图 S3 及补充表 S7、S8,在线补充材料)。在这些预测的蛋白质编码基因中,93.96% 能够通过至少一个公共蛋白质相关数据库(Pfam、NR、InterPro、KEGG、Swiss-Prot、KOG、COG、TrEMBL 和 GO)得到注释(补充表 S9,在线补充材料)。此外,我们还预测到 5,322 个非编码 RNA,包括核糖体 RNA、转运 RNA、微小 RNA 和小核 RNA(补充表 S10,在线补充材料)。
种群遗传分析揭示进化历史
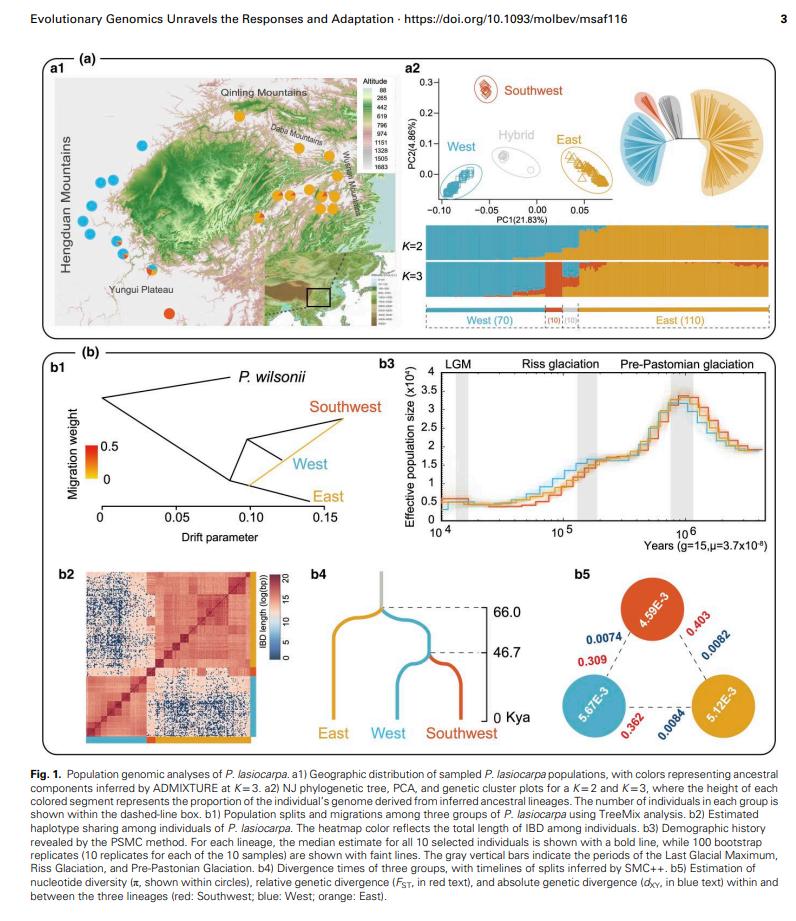
为探究大叶杨种群间的关系及分化情况,我们对分布于中国西南地区其自然分布范围内的 22 个种群的 200 个个体进行了重测序(图 1a1)。测序平均深度约为 25×,比对覆盖率达 95%(补充表 S11,在线补充材料)。经过一系列严格的过滤步骤(见材料与方法),我们共鉴定出 10,567,146 个高质量 SNP、1,534,259 个 indel 和 64,954 个 SV。我们使用随机抽样的 156,324 个经过连锁不平衡(LD)修剪的 SNP 和 9,989 个 SV 来推断种群结构。两个数据集的结果高度一致(图 1a2;补充图 S4,在线补充材料)。
邻接(NJ)系统发育树和主成分分析(PCA)均显示大叶杨存在四个遗传集群,分别对应西部群、东部群、西南部群和混合群(图 1a2)。西部群由来自 8 个种群的 70 个个体组成,主要分布在四川盆地西部的横断山区。东部群包含来自 12 个种群的 110 个个体,分布在四川盆地东部的大巴山和巫山。与西部和东部这两个主要种群群相比,西南部群和混合群各仅包含 10 个个体。此外,ADMIXTURE 聚类分析显示 K=3 时支持度最高,这支持将种群分为西部群、东部群和西南部群(图 1a2)。由于混合群的种群间混杂程度较高,我们在后续分析中排除了该群体。
为进一步深入研究分化和种群历史,我们首先使用 TreeMix 推断三个主要群体间的系统发育树拓扑结构。结果表明,西南部群和西部群拥有较近的共同祖先,且存在一条从东部群到西南部群的迁移边,提示存在基因流(图 1b1)。另一项单倍型共享分析显示,同一群体内的个体比不同群体的个体表现出更多、更长的同源相同(IBD)块。值得注意的是,西部群和东部群之间的单倍型共享模式比它们各自与西南部群之间的更少且更短(图 1b2)。
随后,我们应用成对顺序马尔可夫 coalescent(PSMC)模型来推断每个群体随时间的有效种群大小(Ne)波动情况。结果显示这些群体的种群数量均有共同的下降趋势(图 1b3)。考虑到 PSMC 在推断近期 Ne 轨迹方面的能力有限,我们使用 SMC++ 来探究这些种群更近的历史动态。与西南部群观察到的 Ne 持续减少相比,西部群和东部群在末次盛冰期之后经历了一次共同的瓶颈效应,随后出现了近期的种群扩张(补充图 S5,在线补充材料)。
我们进一步估算了种群群之间的分化时间,结果显示西部群与东部群大约在 66 千年前(Kya)发生分化。西南部群与西部群随后的分化时间估计约为 47 千年前(图 1b4)。
此外,我们调查并比较了种群间的核苷酸多样性(π)以及遗传分化(FST 和 dXY)的模式和水平。与西南部群观察到的较慢的 LD 衰减速率一致(补充图 S6,在线补充材料),其核苷酸多样性也是最低的(π=4.59×10−3),低于西部群(π=5.67×10−3)和东部群(π=5.12×10−3)(图 1b5),这与该群体中观察到的相应瓶颈效应相符。最后,在调整了每个群体的个体数量后,我们从三个群体中随机选择 10 个个体,在 π、LD、FST、dXY 和近期种群历史的分析中获得了高度一致的结果(补充图 S7,在线补充材料)。
全基因组范围内的分化选择与平衡选择信号
在种群分化过程中,古老的多态性可能通过长期平衡选择得以维持,从而在新形成的种群内部及种群之间产生遗传多样性增加的基因组区域(Charlesworth,2006)。此外,祖先的平衡多态性可能通过选择和漂变在后代谱系中进行不均等分配,进而形成分化程度更高的基因组区域(Guerrero 和 Hahn,2017;Han 等,2017)。这些进化力量会导致基因组区域出现异质性的亲缘关系模式,可能会干扰整体的种群结构。为揭示塑造基因组分化格局的潜在进化过程,鉴于西南部群的个体数量少于其他两个主要群体,我们首先将其排除。随后,我们对全基因组上 250 个 SNP 组成的非重叠滑动窗口(基于连锁不平衡衰减情况选择)进行局部主成分分析,并使用多维标度法(MDS;Li 和 Ralph,2019)总结窗口之间的差异。为识别那些遗传变异模式与全基因组种群结构存在差异的基因组区域,我们选取 MDS1 中前 0.5% 和后 0.5% 的窗口,并将其归类为异常区域(图 2a)。与基因组背景窗口相比,这两个极端窗口呈现出不同的系统发育聚类拓扑结构和种群间关系(图 2b)。具体而言,后 0.5% 的窗口显示出更大的种群分离度,而前 0.5% 的窗口则表现出较弱的种群结构,这可能分别反映了分化选择和平衡选择对这些区域的作用。
为进一步探究并确定驱动这些区域基因组变异分化模式的潜在选择因素和过程,我们比较了三种类型窗口的核苷酸多样性水平(π)、Tajima’s D、相对(FST)和绝对(dXY)遗传分化以及跨种群复合似然比统计量(XP-CLR)。与长期平衡选择的效应一致,我们发现前 0.5% 的窗口与基因组背景窗口相比,π 显著升高,Tajima’s D 值更正,dXY 更高,FST 和 XP-CLR 值更低。因此,我们将这些窗口确定为可能受到平衡选择的候选区域(BLS;185 个候选 BLS 窗口,总长度为 1.32 Mb,约占组装基因组的 0.32%)(图 2;补充表 S12,在线补充材料)。为具体验证这些发现,我们应用 β 统计量(β)(Siewert 和 Voight,2017)来检测全基因组范围内长期平衡选择的信号。候选 BLS 区域的 β 得分显著高于邻近区域,为其鉴定提供了强有力的额外支持(补充图 S8,在线补充材料)。此外,对这些 BLS 区域内 166 个候选基因的基因本体论(GO)功能富集分析显示,与代谢过程和生物胁迫响应相关的类别显著富集(补充图 S10b,在线补充材料),包括硫胺素、恶唑啉和噻唑啉的代谢及生物合成过程,这些过程在植物应对 DNA 损伤和病原体攻击中起着关键作用(Anzano 等,2022)。多个受平衡选择的基因与拟南芥中参与植物免疫反应的基因同源(补充表 S12,在线补充材料)。例如,Polas34695 与 ARAF1 同源(图 2d1),ARAF1 编码 α-L – 阿拉伯呋喃糖苷酶,这种酶与植物细胞壁重塑密切相关(Chávez Montes 等,2008)。这种重塑在调节植物免疫反应过程中,对于平衡抗病性和植物发育至关重要(Molina 等,2021)。
相反,后 0.5% 的窗口与背景窗口相比,π 显著降低,Tajima’s D 值更负,dXY、FST 和 XP-CLR 显著更高。因此,这些窗口被确定为可能受到分化选择(DS)的候选区域(185 个候选 DS 窗口,总长度为 1.76 Mb,占组装基因组的 0.42%)(图 2 及补充表 S13,在线补充材料)。对 DS 区域内 163 个候选基因的 GO 富集分析显示,与异生物质刺激和氧化应激反应相关的生物学过程显著富集(补充图 S10a,在线补充材料)。值得注意的是,许多表现出高分化和正选择信号的基因在功能上与器官发育、节律控制和缺氧适应相关(补充图 S9、S10c 及表 S13,在线补充材料)。例如,Polas12612(图 2d2)与拟南芥的 CKX5 同源,已发现该基因调控生殖分生组织的活性和发育(Bartrina 等,2011);此外,Polas20885 与拟南芥的 TIC 同源,经鉴定参与植物昼夜节律的控制(Ding 等,2007)。
环境适应的基因组特征
为识别与各种环境变量相关的基因组变异,我们结合潜在因子混合模型(LFMM;Frichot 等,2013)和全基因组高效混合模型关联分析(GEMMA;Zhou 和 Stephens,2012),在考虑种群结构混杂效应的同时,测试了变异基因型与 29 个环境变量中每个变量的关联。从 LFMM 和 GEMMA 的重叠结果中,我们共识别出 19,955 个推定的适应性变异(补充表 S14,在线补充材料),这些变异广泛分布于全基因组(补充图 S11,在线补充材料)。这些变异不仅包括 SNP 和短 Indel(补充图 S12,在线补充材料),还包括常被忽视的 SV(补充图 S13,在线补充材料)。对 2,044 个带有与环境适应相关变异的基因进行功能富集分析,结果显示与分解代谢和代谢过程相关的 GO 类别显著富集(补充表 S15,在线补充材料),而这些过程对于面临环境变化的植物生存至关重要(Vanholme 等,2010;Pascual 等,2016)。
为验证我们所识别的候选适应性变异的分布模式是由种群间的环境差异驱动,而非仅仅由中性过程或空间结构导致,我们首先进行了 Mantel 检验和偏 Mantel 检验,以评估与环境相关的 “适应性” 变异和随机选择的 “中性” 变异的距离隔离(IBD)和环境隔离(IBE)模式。结果显示,适应性变异和中性变异均表现出强烈的 IBD 模式。然而,通过偏 Mantel 检验控制地理效应后,中性变异的成对遗传距离(FST/1−FST)与环境距离之间仅存在微弱且不显著的相关性(补充图 S14,在线补充材料)。相反,我们观察到适应性变异存在显著的 IBE 模式,这支持了所观察到的遗传 – 环境关联并非简单地受空间自相关混淆的结论。此外,我们使用机器学习算法 —— 梯度森林(GF),比较了候选适应性变异与随机抽样得到的潜在中性变异沿环境梯度的等位基因频率变化。分析显示,与中性参考数据集相比,候选适应性变异的遗传组成变化更大、更迅速,这表明与中性基因座相比,所识别的适应性变异的等位基因频率变异中有更大比例可由相关环境梯度解释(补充图 S12d 和 S13e,在线补充材料)。尽管如此,在识别与局部环境适应相关的变异时仍需谨慎。首先,所使用的大量相关环境变量可能在一定程度上降低多重检验校正的效力,从而提高整体假发现率(FDR)。其次,大叶杨西部和东部种群的独特分布可能无意中导致种群结构与环境适应之间的自相关,使我们对研究结果的解释变得复杂。
单倍型块可能促进局部环境适应
大型单倍型块,尤其是倒位,被认为在局部适应中发挥关键作用,主要是通过抑制共遗传单倍型块内的重组,从而促进多个局部适应性等位基因的捕获和连锁(Kirkpatrick 和 Barton,2006;Savolainen 等,2013;Wellenreuther 和 Bernatchez,2018)。局部种群结构分析方法已被证明是检测多种物种中潜在倒位区域的有力工具(Li 和 Ralph,2019;Todesco 等,2020)。我们使用上述相同的局部 PCA 方法来检测不同的种群结构模式,旨在识别预期为单倍型块的异常区域,这些区域的特征是具有三个群体的清晰聚类模式,且带有两个不同的等位基因(Huang 等,2020)。与之前的分析不同,本分析对每条染色体采用 60 个 SNP 宽的窗口(平均 4,642 bp),以捕获更小、更精确的区域,这些区域在后续分析中可被归为单倍型块。在整合额外的过滤策略以精确定位推定的倒位区域(见材料与方法)后,我们共识别出 218 个候选单倍型块区域,大小从 10 到 650 kbp 不等,包含 647 个基因。这些区域在第一主成分上显示出个体明显聚类为三个群体,对应三种可能的倒位基因型。中间聚类显示出最高的杂合性,与倒位杂合子一致。这些单倍型块内的 GO 类别在乙烯激活的信号转导、磷酸传递信号转导和信号通路中显著富集(补充表 S16,在线补充材料)。
为进一步证实这些单倍型块与结构变异(尤其是倒位)的关联,我们分析了种群规模的重组率、有害突变负荷(0 倍与 4 倍变异的比率)、种群间的遗传分化(FST)以及整体核苷酸多样性(π),并与相同大小的随机抽样基因组区域进行比较(图 3a)。首先,与抑制单倍型间重组的倒位多态性一致,单倍型块区域的重组率显著低于随机抽样的基因组区域。其次,这些单倍型块区域的有害突变负荷高于基因组的其他部分(图 3a),这与由于重组受抑制导致倒位区域的希尔 – 罗伯逊干扰增强一致(McVean 和 Charlesworth,2000)。最后,与其他基因组区域相比,单倍型块区域在种群间表现出显著更高的核苷酸多样性和遗传分化(图 3a)。尽管此处识别的单倍型块表现出类似倒位的特征,但需要注意的是,还需要进一步的独立证据,如长读长测序和比较遗传作图,来确认其结构性质。
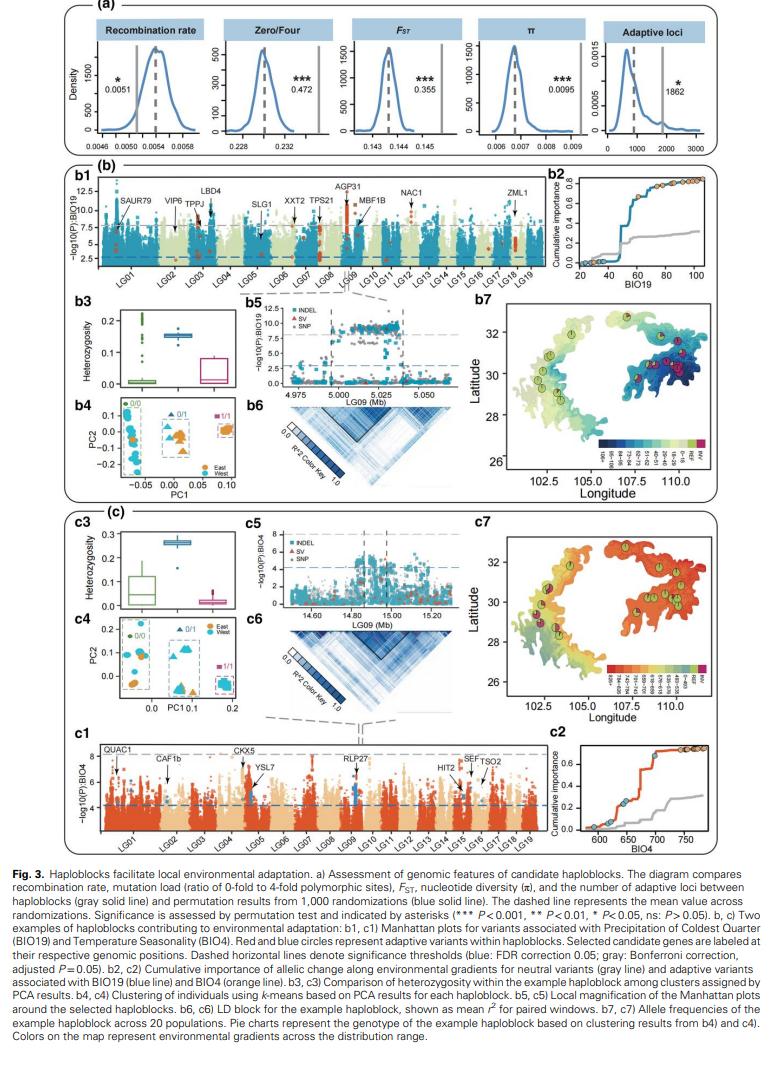
为进一步探究这些单倍型块在大叶杨局部环境适应中的潜在作用,我们比较了单倍型块区域内与相同大小的随机抽样参考区域内环境适应性变异的数量。分析显示,单倍型块区域中存在 1,862 个推定的适应性变异,显著超出预期(图 3a)。此外,我们发现单倍型块区域约占西部和东部种群间已识别的∼1.21 Mb 分化选择区域的 0.55 Mb(31.25%)(补充图 S15a,在线补充材料)。单倍型块区域与分化选择区域的重叠显著高于预期(补充图 S15b,在线补充材料)。这些结果表明,这些单倍型块,特别是推定的倒位多态性,可能作为遗传变异的重要来源,促进局部种群分化和环境适应(Todesco 等,2020;Schaal 等,2022)。例如,9 号染色体上一个具有高连锁不平衡的单倍型块,长度为 44,913 bp,与 508 个候选适应性变异(447 个 SNP、60 个 Indel 和 1 个 SV)相关,这些变异与最冷月降水量(BIO19)密切相关(图 3b)。PCA 显示该区域内存在三个不同的基因型聚类,中间聚类的杂合性更高。这种模式与倒位多态性的预期一致,其中两个极端聚类代表两个不同单倍型的纯合基因型,中间聚类代表杂合子(图 3b)。值得注意的是,该区域内识别出 4 个基因,包括 Polas22852,其与拟南芥的 AGP31 同源,已知该基因参与防御反应期间的维管组织功能,包括对茉莉酸甲酯和脱落酸处理的反应(Liu 和 Mehdy,2007)。另一个值得注意的例子是一个长度为 21,893 bp 的推定适应性倒位,包含 43 个与温度季节性(BIO4)相关的变异(38 个 SNP、4 个 Indel 和 1 个 SV)(图 3c)。该区域内有 3 个基因,包括 Polas23357,其同源基因(RLP27)被发现参与信号转导和防御反应(Wang 等,2008)。
可塑性及基因表达对环境胁迫分化响应的进化分析
除了受选择作用的遗传变化外,我们进一步研究了对环境变化的即时可塑性响应的潜在作用。鉴于基因表达变化对环境波动高度敏感,且常作为基因组与表型之间的桥梁,我们将基因表达水平作为主要性状,探究了基因表达在应对即时环境变化时的可塑性程度。为此,我们分析了来自西部和东部种群的植株在对照和胁迫(高温和水涝)条件下的基因表达差异。这些种群代表了两个主要的遗传集群,所处环境的温度和降水相关条件差异显著,其中东部种群所在地区的温度和降水量高于西部种群(图 4a)。
我们对东部和西部遗传集群的叶片和根组织在对照和胁迫(高温和水涝)两种条件下进行了转录组测序,以模拟未来可能出现的环境胁迫(补充图 S16,在线补充材料)。通过对表达谱的层次聚类初步分析发现,叶片和根组织样本聚成两个不同的组(补充图 S17a,在线补充材料)。主成分分析(PCA)证实了这一发现,其中第一主成分(PC1)解释了 41.85% 的变异,反映了组织特异性差异;第二主成分(PC2)解释了 12.92% 的变异,代表了与处理类型相关的差异(补充图 S17b,在线补充材料)。
为进一步研究和比较两个集群对环境胁迫的转录响应,我们首先分别在叶片和根组织中,对每个集群内的处理组和对照组进行了成对对比(补充图 S18a,在线补充材料)。分析显示,西部和东部集群共享 14% 至 45% 的环境胁迫诱导的可塑性响应基因,重叠程度因具体环境条件而异(补充图 S18b,在线补充材料)。正如预期,高温胁迫诱导的差异表达基因(DEG)富集在与热响应、氧化应激响应和有机氮代谢过程相关的 GO 术语中。同样,水涝胁迫诱导的差异表达基因富集在与系统获得性抗性调控、苯丙烷类代谢过程和防御反应调控相关的 GO 术语中(补充图 S18c,在线补充材料)。
为进一步研究和表征由环境诱导的可塑性响应、遗传集群间的组成型表达差异及其相互作用导致的基因表达变异,我们首先将差异表达基因分为两组:(i)可塑性响应基因,分别比较西部和东部集群在胁迫与对照条件下的差异;(ii)分化表达基因,比较西部和东部集群在对照和胁迫条件下的差异(图 4b1 和 c1)。然后,我们比较了仅与环境条件相关的差异表达基因(可塑性)、仅与遗传集群相关的差异表达基因(分化性)以及同时属于这两个类别的差异表达基因(可塑性和分化性)的数量(图 4b2 和 c2)。在高温和水涝胁迫实验中,以及在叶片和根组织中,结果均显示,在塑造表达差异方面,环境可塑性响应涉及的基因比遗传集群分化或遗传集群依赖性可塑性响应更多(图 4b2 和 c2;补充图 S19A2、B2,在线补充材料)。在高温胁迫下,对环境胁迫表现出独特可塑性响应的基因主要富集在与光合作用、温度刺激响应和次级代谢过程相关的 GO 术语中;在水涝胁迫下,则富集在与系统获得性抗性以及细胞壁组织或生物发生相关的 GO 术语中(补充表 S17,在线补充材料)。相比之下,在任何条件下,遗传集群间表现出分化表达的基因均未显示出显著的 GO 术语富集。同时,在高温胁迫下,表现出遗传集群与环境条件相互作用的基因富集在与谷胱甘肽代谢过程、氧化应激响应和刺激响应相关的 GO 术语中;在水涝胁迫下,则富集在与种子萌发调控以及几丁质代谢和分解代谢过程相关的 GO 术语中(补充表 S18,在线补充材料)。

我们进一步研究了基因表达差异与序列分化之间是否存在关系,以及这种关系在不同类别的可塑性或分化表达基因中是否存在差异。为此,我们估算并比较了西部和东部种群在四组基因中的相对(FST)和绝对(dXY)遗传分化:表现出可塑性表达的基因、组成型分化表达的基因、基于相互作用的表达(可塑性和分化性)的基因,以及在任何条件下均未表现出差异表达的背景基因。与背景基因相比,在高温和水涝胁迫下,可塑性响应基因在 FST 或 dXY 上均未表现出或仅表现出微弱的显著差异(图 4b3、b4、c3、c4)。相反,在叶片组织中,集群间唯一表现出组成型分化表达的基因具有显著更高的 FST 和 dXY(图 4b3、b4、c3、c4),尽管在两种胁迫条件下,根组织中的这种模式较弱(补充图 S19A3、A4、B3 和 B4,在线补充材料)。同时,与背景基因相比,基于相互作用的表达分化基因在 FST 上通常没有显著差异,但在西部和东部种群间的 dXY 值显著更高(图 4b3、b4、c3、c4)。总体而言,这些结果表明,环境诱导的可塑性表达变化与序列进化之间的关联较弱,但强调了遗传集群间的序列分化与组成型差异表达之间存在关联。
种群层面应对未来气候变化脆弱性的进化基因组预测
加速的气候变化正对全球和区域生物多样性构成严重威胁,尤其在那些特有物种丰富、物种多样性极高的山区。四川盆地周边山区经历过多次剧烈的气候波动(He 等,2019)。因此,本研究为深入理解这一关键区域内物种对未来气候变化的响应提供了宝贵机会。为探究适应性变异的分布格局,我们使用经过连锁不平衡修剪的环境关联适应性变异和 7 个成对相关系数 | r|<0.7 的环境变量(BIO1、BIO4、BIO7、BIO8、BIO10、BIO15 和 BIO18)进行了冗余分析(RDA)。通过 RDA,我们计算了适应性指数,该指数根据每个地点的环境预测因子值,量化了景观中所有像素的遗传相似性和差异(Capblancq 和 Forester,2021)。分析显示,大多数适应性变异集中在 RDA1 轴上,该轴解释了 78.83% 的变异。将与 RDA1 相关的适应性梯度外推到景观中,揭示出适应性指数的显著差异:东部地区以较高的年平均温度和最暖季度较高的降水量为特征(RDA1 正得分),而西部地区则表现出明显的降水季节性变化(RDA1 负得分;图 5a)。
接下来,我们研究了自然种群应对未来气候变化的能力和脆弱性,旨在预测哪些种群或区域可能面临最高风险。为此,我们使用梯度森林(GF)方法量化基因组偏移,通过模拟适应性遗传组成对未来气候预测的变化,评估种群为跟上未来环境条件所需的遗传变化程度(Gougherty 等,2021)。除了计算假设原地耐受的局部遗传偏移外,我们还进一步估算了另外两个指标 —— 正向和反向遗传偏移,以在同时考虑原地适应和迁移对未来气候变化的贡献时,评估种群的适应不良情况。正向遗传偏移计算假设种群可以迁移(或重新安置)到欧亚大陆任何地方时的最小预测偏移,而反向遗传偏移计算未来气候中的假设种群与现有分布范围内当前种群之间的最小偏移。
为了衡量不同时间点各种群适应新气候条件所需的遗传变化,我们整合了三种不同未来气候模型(CMCC-ESM2、GISS-E2-1-G、MPI-ESM1-2-HR)在中等(SSP245)和严重(SSP585)共享社会经济路径下,对 2080 年(2061-2080 年)和 2100 年(2081-2100 年)的预测,以考虑气候模型之间的变异性。不同模型的局部、正向和反向偏移估计结果高度一致(补充图 S20,在线补充材料),表明西部地区的种群比分布范围东部地区的种群表现出更大的遗传偏移。此外,无论是使用所有气候变量还是从 RDA 分析中识别的 7 个不相关变量,基因组偏移的空间模式都密切一致(补充图 S21,在线补充材料)。因此,在正文中,我们展示了在 2060-2080 年期间,中等情景(SSP245)下使用所有气候变量的三个气候模型的平均结果(图 5b)。同样,预测的局部、正向和反向偏移显著较高的模式表明,分布在横断山区的西部种群在未来气候下可能特别脆弱,因为在该地区,无论是原地适应还是迁移都不太可能有效降低种群层面对气候变化的脆弱性。
最后,为了进一步评估脆弱的西部种群是否比东部种群积累了更高的遗传负荷(Robinson 等,2023),我们通过计算每个种群的衍生功能丧失(LoF)和有害变异相对于同义变异的比例来衡量基因组负荷。我们的研究结果显示,西部和东部种群之间的 LoF 变异负荷没有显著差异,这意味着两组中的选择都能有效清除严重有害的突变。然而,我们观察到,在采样种群中,有害变异与同义变异的比率与预测的局部基因组偏移之间存在显著的正相关关系(图 5c)。与较高的遗传偏移一致,西部种群在清除中度有害变异方面的效率较低,这凸显了对这些脆弱种群加大保护力度和关注度的必要性(图 6)。
讨论
全球气候变化正日益加剧生物多样性丧失。为更好地理解和预测物种在气候变化面前的响应与动态,必须采用整合遗传和可塑性效应的进化视角(Waldvogel 等,2020;Sang 等,2022)。对当前及未来气候变化的未来进化响应预测,在很大程度上依赖于对历史和当代适应性进化响应的研究(Martin 等,2023;Sandercock 等,2024)。在山区,高度异质的环境和地理屏障往往会推动物种的种内遗传分化和局部生态适应(He 等,2019)。本研究以分布于东南亚山区生物多样性热点地区的特有优势树种 —— 大叶杨(P. lasiocarpa)为例,凸显了整合进化基因组学方法在预测和监测物种及种群对全球气候变化响应方面的独特价值。通过利用新组装的高质量参考基因组和种群基因组数据集,我们探究了自然选择、环境适应和基因表达可塑性在历史、当代和未来时间尺度上对大叶杨种群气候变化响应的复杂且相互交织的作用(图 6)。

我们的研究结果首先揭示了西部和东部种群之间存在明显的遗传分化,四川盆地既是地理屏障也是遗传屏障,导致大叶杨形成环状分化模式。两个谱系均表现出一致的种群历史,表明第四纪的气候波动对两组种群产生了相似的影响。除了遗传漂变和种群动态等中性进化过程外,我们的研究结果还表明,分化选择(DS)和长期平衡选择在塑造谱系内部和谱系之间的基因组变异模式中发挥了关键作用。分化选择区域在两个谱系中均具有强烈的正选择信号,种群间的相对和绝对遗传分化(FST 和 dXY)水平较高。这些区域还呈现出能清晰区分西部和东部谱系的系统发育拓扑结构。值得注意的是,这些区域内的基因富集在与环境刺激和应激反应相关的类别中,强调了它们在驱动两个独立谱系对当地气候的差异适应方面的潜在作用(Nosil 等,2009)。相反,我们在西部和东部种群中均识别出具有显著遗传多样性和高水平多态性的区域,这些区域可能由长期平衡选择维持(Charlesworth,2006)。这些区域显著富集了参与代谢过程和生物胁迫响应的基因。总之,这些发现凸显了分化选择和长期平衡选择,以及历史种群过程在塑造现有遗传变异以适应气候变化方面的重要作用。
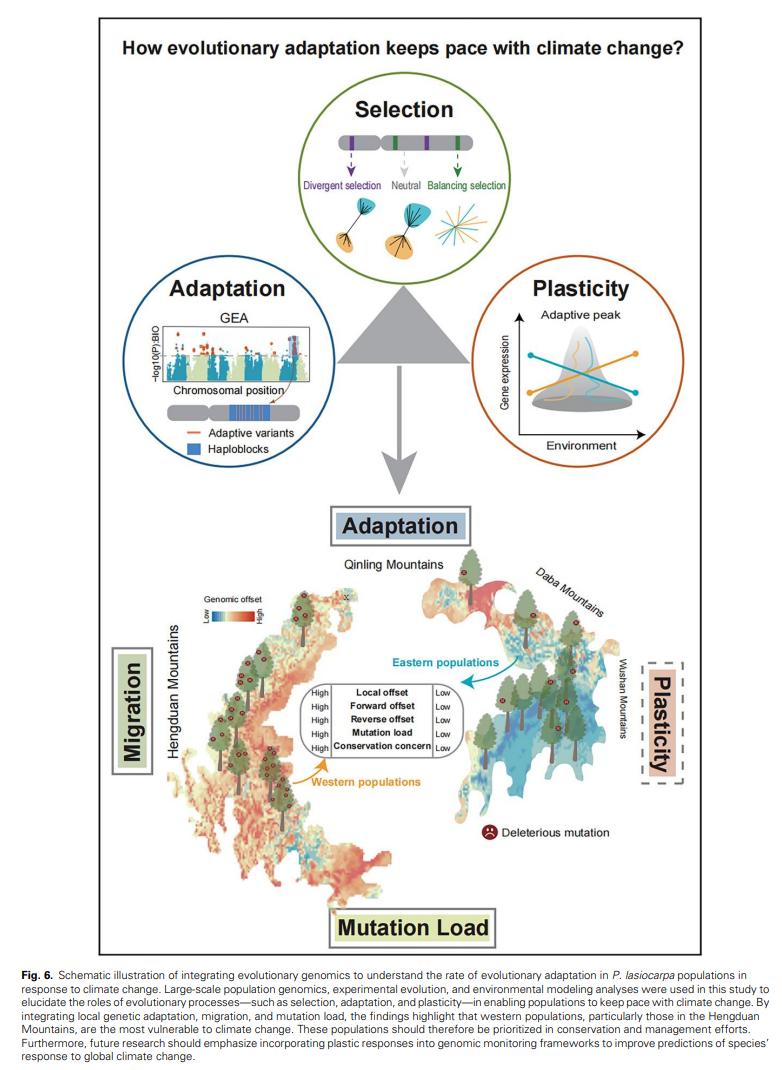
识别与气候相关的适应性遗传变异并描述其空间分布模式,可为面临气候变化的种群的适应潜力提供宝贵见解(Bernatchez 等,2024)。我们的研究结果揭示了众多多态性单倍型块,其特征与倒位一致,且显著富集了局部气候适应的候选变异。与最近在向日葵(Todesco 等,2020)、鹿鼠(Harringmeyer 和 Hoekstra,2022)和入侵杂草物种(Battlay 等,2023)中的研究一致,我们的结果表明,单倍型块多态性可能也与大叶杨自然种群的局部环境适应密切相关。这一点尤为重要,因为这些区域内重组的抑制可以保护局部有利的等位基因组合免受迁移的稀释,尤其是在高基因流的情况下(Schaal 等,2022)。然而,目前尚不清楚此处识别的单倍型块是否与倒位相关,或者是否代表经历低重组的紧密连锁等位基因。未来涉及种群水平长读长测序和分子实验的研究对于更好地描述这些单倍型块至关重要。此外,我们观察到单倍型块区域往往会积累有害突变,这可能是由于重组受抑制导致净化选择效率降低所致(McVean 和 Charlesworth,2000)。这就提出了一个尚未解决的问题,即在单倍型块内,集中气候适应性变异的益处与积累有害突变的代价之间如何权衡。解决这一矛盾需要进一步研究。
此外,与对气候变化的遗传响应相比,分子表型可塑性为应对气候变化的负面影响提供了即时缓冲(Sandoval-Castillo 等,2020;Campbell-Staton 等,2021;Huang 等,2023)。我们的研究结果揭示了众多对环境波动表现出转录可塑性的基因。环境诱导的可塑性基因表达变化比遗传集群间的组成型差异更为普遍,一些差异表达基因(DEG)在环境条件和遗传集群中表现出可塑性和组成型分化表达的交互模式。在研究表达分化与序列进化的关系时,与背景基因相比,可塑性响应基因在集群间的遗传分化极小。相反,组成型分化表达基因的相对和绝对遗传分化值均显著高于背景基因。这些结果表明,西部和东部谱系中这些基因内部或附近的突变积累可能与基因表达的进化变化相关(Wos 等,2021;Szukala 等,2023)。然而,将组成型差异表达与其基因组基础(如顺式作用变异)联系起来的潜在分子机制仍有待进一步研究(Jacobs 等,2020;Ballinger 等,2023;Durkin 等,2024)。此外,分化选择在驱动大叶杨西部和东部种群分化过程中是否对适应性基因表达进化起到作用,目前尚不清楚(Bhaskara 等,2023)。可塑性响应在多大程度上驱动转录组水平的进化变化并促进对未来极端气候的适应,也在很大程度上未知(Ghalambor 等,2015;Campbell-Staton 等,2021)。因此,需要进一步研究以确定短暂和新兴的可塑性响应是否以及如何成为遗传适应新的快速变化环境的垫脚石(Merilä 和 Hendry,2014;Vinton 等,2022;Coates 等,2025)。
最后,探究和理解当代气候适应背后的进化力量和基因组结构,为预测物种对当前和未来气候变化的响应提供了关键基线(Aguirre-Liguori 等,2021;Bernatchez 等,2024)。通过考虑种内适应性变异和迁移的缓解效应,我们的研究结果表明,西部种群对未来气候变化特别脆弱,因为与东部种群相比,它们表现出更高的局部、正向和反向基因组偏移。此外,我们发现西部种群携带的轻度有害突变的遗传负荷比东部种群更大。这表明过去的历史种群统计和种群动态可能加剧了这些种群的衰退,削弱了净化选择,并降低了它们在环境条件变化时的适合度。值得注意的是,西部种群主要分布在横断山区,该地区以高度异质的环境和显著的生物多样性著称,包括许多特有山地物种(Myers 等,2000)。如果这些关键林木物种对未来气候变化的脆弱性日益增加,可能会进一步危害当地生态系统,并威胁该地区的整个特有动植物群。为减轻局部适应不良,从东部向西部种群进行辅助基因流可能是一种有效的策略,特别是考虑到分隔这些种群的四川盆地形成了天然的遗传屏障(Aitken 和 Whitlock,2013)。引入预先适应更温暖湿润气候的东部种群的有利基因型,可能会增强西部种群应对新气候条件的适应能力。
总之,我们的研究整合了各种进化过程 —— 包括种群历史、选择压力、局部环境适应、基因表达可塑性和遗传负荷 —— 以全面理解种群水平对气候变化的脆弱性。尽管如此,仍需要精心设计的公共花园和移植实验来验证大叶杨中观察到的环境适应模式和未来气候变化下的基因组预测。这一点尤为重要,因为多种进化过程无法完全区分,特别是考虑到该物种明显的西部和东部分布模式(Lind 等,2024;Lotterhos,2024)。此外,环境诱导的可塑性在塑造基因表达变异和分化中的重要作用,强调了研究可塑性、遗传变异、选择和适应之间相互作用的必要性。具体而言,理解遗传变化、即时可塑性响应及其相互作用的相对贡献,对于揭示适应、脆弱性和恢复力的潜在机制至关重要。未来的研究还应优先将可塑性响应纳入基因组监测和预测框架,以改进对物种对全球气候变化响应的预测。
材料与方法
基因组测序与组装
采集位于中国四川省眉山市新桥镇(东经 103°51′36″,北纬 30°9′36″)的一株野生大叶杨新鲜幼叶,并用液氮冷冻。采用 CTAB 法(Rogers 和 Bendich,1985)从叶片中提取高质量基因组 DNA。对于长读长测序,构建了 DNA 片段>20 Kbp 的文库,并使用 Oxford Nanopore Technologies 的 PromethION 平台进行测序。对于短读长测序,构建了插入片段大小为 350 bp 的 150 bp 双末端文库,并在 Illumina HiSeq X Ten 平台上测序。为实现染色体级别的基因组组装,构建了 Hi-C 测序文库,用 DpnⅡ 限制酶处理后,在 Illumina NovaSeq 平台上测序。
我们按以下步骤进行从头组装:首先,对长测序读长进行自我校正后,使用 NextDenovo(
https://github.com/Nextomics/NextDenovo)组装成重叠群。为获得高质量组装结果,使用 Racon(Vaser 等,2017)用 Nanopore 读长进行三轮 polishing,并用 Nextpolish v1.0.5(Hu 等,2020)用 Illumina 读长进行四轮 polishing。然后,使用 purge_haplotigs v1.1.1,通过设置三个读长深度阈值(低阈值 5、“单倍体” 与 “二倍体” 峰中点 66、高阈值 170),从所得重叠群水平组装结果中去除杂合重叠群(Roach 等,2018)。接下来,用 fastp v0.20.0(Chen 等,2018)过滤 Hi-C 读长,并使用 bowtie2 v2.3.2(Langmead 和 Salzberg,2012)将其比对到重叠群水平组装结果上。然后,合并每对唯一比对的读长,使用 LACHESIS(Burton 等,2013)将重叠群聚类、定向并连接成 19 条假染色体。最后,为验证组装的完整性和准确性,我们计算了 BUSCO(Simão 等,2015)得分,并使用 BCFTOOLS v1.11(Li,2011)估计 Illumina 短读长的比对率。
基因预测与功能注释
在基因预测前,使用 EDTA v1.9.3(Ou 等,2019)进行初步转座元件(TE)注释,并使用 TEsorter v1.2.5(Zhang 等,2022)对注释为 “LTR/unknown” 的 TE 进行重新分类。然后,使用 RepeatMasker v4.10(Tempel,2012)用 TE 文库对全基因组序列进行屏蔽。为预测蛋白质编码基因,我们整合了同源预测、RNA-seq 预测和从头预测三种策略。首先,通过 TBLASTN(ncbi-BLAST v2.2.28)(Camacho 等,2009)将六个物种(毛果杨、胡杨、细枝柳、紫柳、拟南芥和葡萄)的蛋白质序列比对到大叶杨基因组,并使用 Genewise v2.4.1(Birney 等,2004)解析所得比对结果进行同源预测。然后,使用 Trinity v2.8.4(Haas 等,2013)通过无参考基因组和有参考基因组两种方法组装叶片和茎组织的 RNA 测序读长得到转录本,再用 PASA v2.4.1(Haas 等,2008)将其比对到参考基因组进行 RNA-seq 预测。此外,利用同源预测和 RNA-seq 数据得到的基因模型,使用 Augustus v3.3.2(Stanke 和 Morgenstern,2005)进行从头基因预测。最后,使用 EvidenceModeler v1.1.1(Haas 等,2008)将上述所有基因模型整合为一个综合基因集,随后通过三轮 PASA v2.4.1 进行优化。
基于 BLAST,以 1e-5 的 E 值阈值,对照 9 个知名公共蛋白质数据库进行基因功能注释:蛋白质家族数据库(Pfam)、NCBI 非冗余蛋白质数据库(NR)、interproscan 数据库、KEGG 数据库、Swiss-Prot 蛋白质数据库、真核生物同源蛋白质组(KOG)、同源蛋白质簇(COG)、欧洲分子生物学实验室翻译数据库(TrEMBL)和 GO 数据库。使用 InterProScan v5.32-71.0(Jones 等,2014)程序,采用默认参数识别基因的推定结构域和 GO 术语。
基因组重测序、读长比对与变异检测
从该物种自然分布范围内的 22 个自然种群中采集了 200 份野生大叶杨材料进行全基因组重测序。每个种群内的采样个体间距至少为 100 m。使用 DNeasy Tissue Kit(QIAGEN)从新鲜成熟叶片中提取基因组 DNA。构建 150 bp 的 Illumina 双末端文库,在 Illumina NovaSeq 6000 平台上测序,目标覆盖度为每个个体至少 20×。
在进行读长比对前,使用 Fastp v0.20.0 对原始测序读长进行修剪和过滤。参数设置为 “-f 5 -F 5 -t 5 -T 5”,从双末端读长的前端和尾部各修剪 5 个碱基。参数 “-n 0 -q 20 -u 20” 用于保留无 N 碱基、Phred 质量值>20 且不合格碱基<20% 的读长。在修剪前后,使用 FastQC v.0.11.7(Davis 等,2013)评估读长质量。然后,使用 BWA-MEM(Li,2013)将剩余的读对比对到我们的高质量参考基因组上,并使用 SAMtools v.1.9(Li 等,2009)按默认参数进行排序。使用 Picard v.2.18.21(
https://broadinstitute.github.io/picard/)中的 MarkDuplicates 工具标记 PCR 重复序列。对于基因组变异识别,使用 Genome Analysis Toolkit(GATK v.3.8.1;DePristo 等,2011)及其子组件 HaplotypeCaller 进行 SNP 和 Indel 检测,以获得每个样本的变异。然后,使用 GenotypeGVCFs 工具进行联合基因分型,选项设为 “-all-sites” 以输出变异位点和非变异位点。使用 VCFtools v0.1.16(Danecek 等,2011)过滤 SNP 和 Indel,保留双等位基因变异,且基因型缺失率<20%(将读长深度(DP)<5 和基因型质量(GQ)<10 的基因型视为缺失)。使用 DELLY v0.8.38(Rausch 等,2012)检测结构变异(SV),保留长度>50 bp、断点精确且基因型缺失率<20% 的变异(将 DP<5 和 GQ<10 的基因型视为缺失)。最后,使用 SNPable(
https://lh3lh3.users.sourceforge.net/snpable.shtml)屏蔽读长无法唯一比对的基因组区域,并进一步过滤这些区域内的变异。
系统发育、混合 ancestry 与种群结构分析
为分析种群结构,仅保留次要等位基因频率(MAF)>5% 的 SNP。使用 PLINK v1.90b6.18(Purcell 等,2007)通过基于连锁不平衡(LD)的 SNP 修剪排除高度相关的 SNP,参数设置为 “–indep-pairwise 50 10 0.2”(在全基因组的 50 个 SNP 滑动窗口中,以 10 个 SNP 为步长,去除与其他 SNP 的相关系数(r²)>0.2 的任何 SNP)。使用 PLINK 和 MEGA-X(Kumar 等,2018)构建邻接(NJ)系统发育树,并在 Figtree v1.4.4(
http://tree.bio.ed.ac.uk/software/figtree/)中可视化。还使用 EIGENSOFT v.7.2.1(Patterson 等,2006)中的 smartpca 程序进行主成分分析(PCA),以进一步评估遗传结构。使用 ADMIXTURE v1.3.0(Alexander 等,2009)检查所有个体的种群结构,预定义的遗传集群(K)数量设置为 1 到 10。通过具有最低交叉验证误差的 K 值确定最适合的祖先群体数量。此外,为评估和验证检测到的 SV 质量,我们使用相同的方法和参数对修剪后的 SV 数据集进行种群结构分析。
遗传多样性、连锁不平衡及种群分化
使用 pixy v1.0.4.beta1 程序(Korunes 和 Samuk,2021),在 100 Kbp 的非重叠窗口中,同时考虑多态性位点和单态性位点,计算组内核苷酸多样性(π)和组间遗传分化(FST 和 dXY)。为评估连锁不平衡衰减,使用 PopLDdecay v3.41(Zhang 等,2019)计算 200 Kbp 窗口内所有次要等位基因频率>0.05 的 SNP 对的平方相关系数(r²)并绘图。为进一步消除计算所用个体数量的影响,我们还从每个组中随机抽取 10 个个体来计算这些参数。
种群历史和分化时间估计
分别使用成对顺序马尔可夫 coalescent 模型 PSMC v0.6.5-r67(Liu 和 Hansen,2017)和 SMC++ v1.15.4(Terhorst 等,2017)估计古老和较近期的种群历史。假设突变率为 3.75×10⁻⁸(每个位点每代的突变数),世代周期为 15 年(Ingvarsson,2008),然后将缩放结果转换为年份和有效种群大小(Ne)。对每组 10 个选定个体进行 PSMC 分析,每个个体进行 10 次 bootstrap,采用默认参数设置。使用 10 个个体的中位数估计值来代表每组的 Ne 波动情况。为推断更近的种群历史,使用 SMC++ 中的 vcf2smc 命令将含高质量多态性 SNP 的 VCF 文件转换为 SMC 格式,然后对每组运行 estimate 命令,采用默认参数。为估计分化时间,对每组配对的 VCF 文件运行 SMC++ 程序中的 vcf2smc 和 split 命令,采用默认参数。
为在系统发育背景下探究基因流模式,使用修剪后的共有 SNP 集,并选择威尔逊杨(与大叶杨同属白杨组)作为外类群。然后,通过 TreeMix v.1.13(Pickrell 和 Pritchard,2012)推断迁移事件,参数设置为 “-bootstrap 5000 -m 0-5(-m 为迁移事件)”。使用 beagle v4.1(Browning 和 Browning,2013)检测所有个体间的 IBD 块,参数设置为 “window=100,000;overlap=10,000;ibdtrim=100;ibdlod=5;ibdcm=1e-5”。
选择作用下基因组区域的识别
为识别可能受到选择且表现出异质亲缘关系模式的异常种群结构基因组区域,使用 R 包 lostruct v0.0.0.9000(Li 和 Ralph,2019)对来自西部和东部种群的 180 个个体进行局部 PCA。根据连锁不平衡衰减情况和捕获有意义选择信号的需求,我们选择 250 个 SNP 的窗口大小进行此分析。首先将大叶杨基因组划分为 40,380 个连续且非重叠的窗口,每个窗口包含 250 个 SNP。去除物理长度超过 20 Kbp 的异常大窗口,因为这些窗口可能受到高比例重复序列的影响。为测量不同窗口间个体亲缘关系的相似性,计算欧氏距离矩阵并通过 MDS 转换进行可视化。将具有极端 MDS1 值(MDS1 值的前 0.5% 和后 0.5%)的窗口定义为异常窗口。我们进一步分别对顶部异常区域、底部异常区域和背景区域进行 PCA 并构建 NJ 系统发育树。
由于这两组异常窗口表现出与分化选择和平衡选择信号一致的系统发育聚类拓扑结构,我们通过将它们与基因组的其余背景部分进行比较,进行以下分析以进一步评估选择作用:(i)使用 pixy v1.0.4.beta1 计算西部和东部种群组内和组间的核苷酸多样性(π)以及遗传分化(FST 和 dXY);(ii)使用 VCFtools 在 10 Kbp 非重叠窗口中分别为西部和东部种群计算 Tajima’s D 统计量;(iii)使用 SweepFinder2(DeGiorgio 等,2016),基于极化 SNP 的全基因组位点频率谱(SFS),在 2 Kbp 非重叠窗口中分别为西部和东部种群计算复合似然比统计量(CLR);(iv)使用 XP-CLR 软件包(Chen 等,2010)在 1 Kbp 非重叠窗口中计算 XP-CLR 得分,以专门检测分化选择信号;(v)基于极化 SNP 的全基因组 SFS,通过 BetaScan2(Siewert 和 Voight,2017)在 1 Kbp 非重叠窗口中计算 β 统计量(β),以专门检测长期平衡选择信号。最后,通过 R 包 gggenes(
https://github.com/wilkox/gggenes)展示候选区域中候选基因的结构。
环境关联遗传变异的检测
为筛选局部适应性环境变异,我们提取并下载了与种群采集地点地理坐标相对应的 29 个环境变量(补充表 S14,在线补充材料)。这些变量包括 1970-2000 年期间的 19 个与温度和降水相关的气候变量以及水汽压(kPa),分辨率为 30 秒,均从 WorldClim 公共数据库(https://www.worldclim.org)获取。此外,从 SoilGrids 网站(https://soilgrids.org)下载了 2017 年 5-15 米深度的 4 个土壤变量,分辨率为 1000 米。从 glUV 数据集(https://www.ufz.de/gluv/)获取 2004-2013 年每月的紫外线(UV)辐射变量,分辨率为 15 角分,随后转换为 5 个变量,包括四个季度和全年的紫外线辐射。
我们保留常见变异(次要等位基因频率>5%),共得到 5,315,353 个 SNP、687,429 个 indel 和 27,980 个 SV,并采用两种方法识别环境关联变异。首先,使用 R 包 LEA v3.2.0(Frichot 和 François,2015)中的 LFMM(LFMM;Frichot 等,2013)搜索等位基因频率与 29 个环境变量中每个变量的显著关联。基于 ADMIXTURE 在 20 个种群中推断的最佳祖先集群数量,本分析使用两个潜在因子来考虑种群结构的影响。对于每个环境变量,LFMM 运行 5 次重复,包括 5,000 次燃烧周期和随后的 10,000 次迭代。计算 5 次运行结果的 z 分数中位数,通过 lambda 重新校准,并使用 FDR 方法对所得 P 值进行多重检验校正,将 5% 的 FDR 设为显著性阈值。此外,我们使用 GEMMA v0.98.3 程序(Zhou 和 Stephens,2012)采用线性混合模型方法进行全基因组环境关联研究,将环境作为表型数据。该方法利用从所有 6,030,762 个变异生成的中心化亲缘关系矩阵(-gk 1)来考虑个体间的相关性和种群结构。与 LFMM 类似,我们应用 FDR<5% 来控制变异数据集的多重检验。
最后,为评估所识别的适应性变异的可靠性,我们评估了等位基因频率沿环境梯度的变化,并比较了所识别的适应性变异与相同数量变异的中性参考集之间的变化模式。按照 Aguirre-Liguori 等(2021)使用的方法,本分析通过 R 包 “gradientForest”(Ellis 等,2012)中实现的机器学习算法进行,以检测等位基因频率(响应变量)沿气候梯度(预测因子)的非线性变化。我们使用参数 “ntree=400,maxLevel=log2 (0.368×n/2),corr.threshold=0.40” 运行 GF,并计算等位基因频率变化的累积重要性作为沿气候变量的分裂重要性之和。通过 R 包 ggplot2 可视化等位基因频率沿环境梯度的变化。此外,为进一步评估地理和环境对适应性和中性变异基因组变异的影响,我们使用线性化的 FST(FST/1−FST),分别通过 Mantel 检验和偏 Mantel 检验研究距离隔离和环境隔离,其中基于所有缩放环境变量的欧氏距离用作环境距离的度量。
单倍型块的表征
为识别可能由单倍型块或倒位导致的具有异常种群结构的基因组区域,我们使用 R 包 “lostruct”(Li 和 Ralph,2019)进行局部主成分分析。计算主成分分析图谱(使用前两个主成分)之间的欧氏距离,以评估 60 个 SNP 宽窗口之间的亲缘关系,确保在噪声和信号之间取得平衡。通过将多维标度得分(MDS)与每条染色体上每个窗口的基因组位置作图,将这些距离可视化。为改进潜在倒位的检测,我们利用了三维 MDS 轴。接下来,我们计算每个窗口沿每条 MDS 轴的绝对 z 分数(标准分数),表示目标窗口的 MDS 值与全基因组平均值之间的距离,以标准差为单位。将至少 10 个相邻窗口的 z 分数>1.5 的基因组区域定义为初步候选单倍型块。
为进一步证明 MDS 异常值可能由单倍型块引起,我们进行了额外分析以确认以下基因组特征:(i)候选区域内所有 SNP 的主成分分析应显示三个不同的聚类,分别对应 0/0、0/1 和 1/1 基因型;(ii)与 0/0 和 1/1 基因型相比,0/1 基因型应表现出最高的杂合性。最后,对于选定的倒位示例,检查并比较单倍型块区域和邻近区域内的连锁不平衡模式。
然后,我们比较了单倍型块区域与相同大小的随机抽样基因组区域之间的 π、种群间 FST、0 倍与 4 倍同义变异的比率、气候适应性基因座的数量以及种群规模的重组率(通过 LDhat 的 Interval 程序计算(McVean 和 Auton,2007),参数设置为 “-its -bpen 5 -samp 2000”)。最后,我们使用 topGO v2.42.0(Alexa 和 Rahnenführer,2009)进行基因本体论富集分析,以确定单倍型块区域中的基因是否富集特定的生物学功能。
胁迫实验设计与 RNA 测序
分别从大叶杨西部(东经 103°7′12″,北纬 28°43′12″)和东部(东经 109°28′48″,北纬 29°59′24″)种群的个体上收集成熟种子。种子首先在培养皿中萌发,然后转移到花盆中,在温室中生长 4 个月,温度条件为白天 25°C、夜间 20°C,光周期为 16 小时光照、8 小时黑暗。
生长结束后,将两个种群的幼苗分为三组:对照组、高温胁迫组和水涝胁迫组,每组包含 3 个生物学重复。对照组保持在 25°C/20°C(白天 / 夜间)的标准条件下。水涝胁迫组的植株被转移到水箱中,完全水涝处理,水位高于土壤表面 3-5 厘米,在 25°C/20°C(白天 / 夜间)条件下持续 15 天。高温胁迫组被置于培养箱中,白天 42°C、夜间 35°C,处理 3 天。处理最后一天,从所有重复的相同位置采集叶片和根组织,迅速在液氮中冷冻,并在 – 80°C 下保存。从这些组织中提取总 RNA,制备 RNA-seq 文库,并在 DNBSEQ-T7 平台上测序,生成 150 bp 的双末端读长。
基因表达分析
使用 Trimmomatic v0.39 对 RNA 读长进行过滤和修剪(采用默认参数)后,我们使用 HISAT v2.2.1(Kim 等,2015)将干净的读长比对到参考基因组,并使用 StringTie(Pertea 等,2015)对基因表达进行定量。然后通过 Python 脚本 prepDE.py 对基因表达值进行标准化,生成读长计数数据。基于此标准化数据,我们首先创建相关矩阵,并在处理样本和重复之间进行主成分分析。去除读长数少于 10 的基因,仅保留表达的基因用于下游分析。
为量化表达变化,我们使用 DESeq2(Love 等,2014)在两个种群的每组中,分别检测叶片和根组织中对照组与胁迫处理个体之间环境诱导的可塑性差异表达。此外,我们还检测了遗传集群的影响,即在叶片和根组织中,识别在对照和胁迫处理条件下西部和东部种群之间组成型分化的差异表达基因。差异表达基因的鉴定采用 FDR<0.05 且 – 2<log2 倍变化>2 的阈值。
我们随后检查并可视化这四组差异表达基因之间的重叠,这些基因随后被分为三类:(i)可塑性响应基因:仅对环境胁迫处理有响应的差异表达基因;(ii)分化响应基因:仅在西部和东部遗传集群种群之间存在分化的差异表达基因;(iii)可塑性和分化响应基因:由于环境处理和遗传集群均表现出差异表达的基因。为进一步探究基因表达分化与序列分化速率之间的关联,我们使用 pixy 程序(v1.0.4.beta1)估算并比较西部(70 个个体)和东部(110 个个体)遗传集群之间每个基因的相对(FST)和绝对(dXY)遗传分化度量。在以下基因类别中进行这些比较:可塑性响应基因、分化响应基因、可塑性和分化响应基因以及其他背景基因。使用 Wilcoxon 秩和检验检验类别之间差异的显著性。
适应性指数
为探究大叶杨的适应性遗传格局,我们使用 R 包 “vegan v2.6-4”(Dixon,2003)进行冗余分析,设置 K=2 以计算适应性指数。仅使用来自基因组环境关联分析的经过连锁不平衡修剪的适应性变异,以及 7 个不相关(成对相关系数 | r|<0.7)的环境变量(即 BIO1、BIO4、BIO7、BIO8、BIO10、BIO15 和 BIO18)。通过 R 包 ggplot2 将适应性指数外推到整个物种分布范围,以可视化适应的地理模式。
基因组偏移评估
对于每个采样地点,从 WorldClim CMIP6 网站下载了两种共享社会经济路径(SSP245 和 SSP585)下 2061-2080 年和 2081-2100 年期间 19 个气候变量的未来 BIOCLIM 数据,分辨率为 2.5 角分。整合了三个气候模型(CMCC-ESM2、GISS-E2-1-G 和 MPI-ESM1-2-HR)的数据。使用机器学习方法 —— 梯度森林(GF)预测对环境变化的基因组偏移。基于 19,955 个适应性变异和 15 个已识别出适应性变异的环境变量,构建梯度森林模型,参数设置为 ntrees=500、maxLevel=log2 (0.368×n/2)、corr.threshold=0.5。
计算了三个潜在适应不良的指标:局部偏移(种群在原地对新气候条件的脆弱性)、反向偏移(如果个体可以重新安置到物种分布范围内的任何地方的潜在适应不良)和正向偏移(假设个体可以迁移到欧亚大陆任何地方的最小适应不良)(Gougherty 等,2021)。这些指标分别可视化为 RGB 图像的红色、蓝色和青绿色波段。此外,我们评估了不同最大允许迁移距离(包括 100、250、500 和 1000 公里以及欧亚大陆范围内无限制)下正向偏移的变化。结果表明,正向偏移随着迁移距离的增加而显著降低,在 500 公里及以上时趋于稳定(补充图 S22,在线补充材料)。因此,呈现了具有无限制扩散距离的正向偏移结果,并将其投影到地图上。
遗传负荷评估
为评估大叶杨种群中的有害遗传负荷,使用 SIFT 4G 注释器(Vaser 等,2016)将变异注释为同义或非同义,并计算非同义变异的 SIFT 分数。然后将变异分为四类:功能丧失(LOF)、有害(SIFT 分数<0.05)、耐受(SIFT 分数>0.05)或同义。此外,以毛果杨为外类群,使用 est-sfs(Keightley 和 Jackson,2018)确定每个 SNP 位置的衍生等位基因与祖先等位基因状态。衍生突变(功能丧失和有害)数量与同义变异数量的比率用作遗传负荷的替代指标。
Zhiqin Long、Yupeng Sang、Jiajun Feng、Xinxin Zhang、Tingting Shi、Lushui Zhang、Kangshan Mao、Loren H. Rieseberg、Jianquan Liu、Jing Wang
Evolutionary Genomics Unravels the Responses and Adaptation to Climate Change in a Key Alpine Forest Tree Species
Molecular Biology and Evolution, 2025, 42, 1–21. https://doi.org/10.1093/molbev/msaf116
文内图片及封面图片来源原文

免责声明:本站所有文章内容,图片,视频等均是来源于用户投稿和互联网及文摘转载整编而成,不代表本站观点,不承担相关法律责任。其著作权各归其原作者或其出版社所有。如发现本站有涉嫌抄袭侵权/违法违规的内容,侵犯到您的权益,请在线联系站长,一经查实,本站将立刻删除。 本文来自网络,若有侵权,请联系删除,如若转载,请注明出处:https://haidsoft.com/185594.html

