大家好,欢迎来到IT知识分享网。
Pymol入门教程–基础
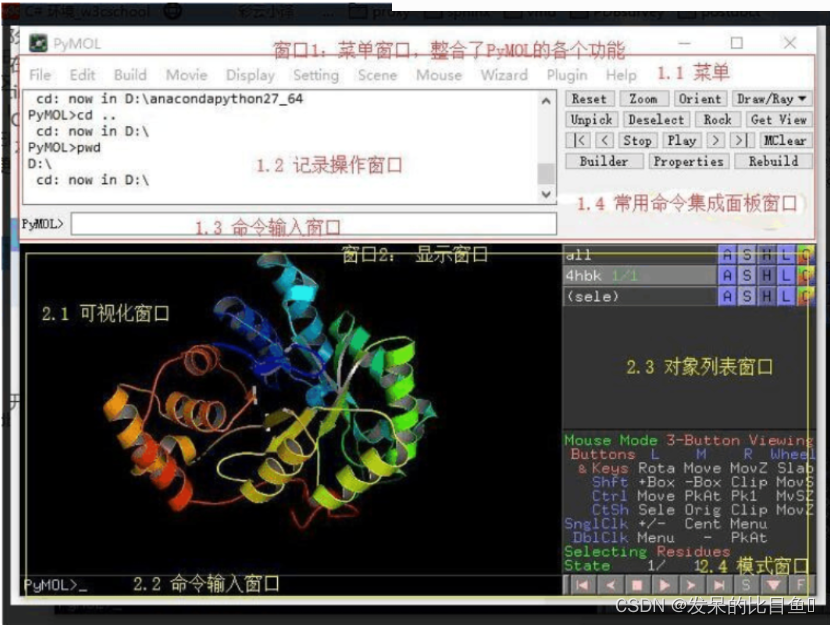
软件界面介绍
内置demo介绍
打开PyMOL,点击1.1菜单窗口的Wizard菜单,然后点击Demo->Representations然后在2.3对象窗口和2.4模式窗口之间会出现各种示例:
- Representations
- Cartoon Ribbons
- Roving Detail
- Roving Density
- Transparency
- Ray Tracing
- Sculpting
- Scripted Animation
- Electrostatics
- CGOs
- Molscript/R3D Input
- End Demonstration 关闭示例
设置和查看工作路径
点击菜单窗口File->Working Directory->Change可以查看现在的工作目录在哪里,也可以设置新的路径作为工作目录。
工作目录的作用:默认文件的打开和保存都是从该文件开始。下载文件的保存位置也是在工作目录。这一点与R语言类似,我也是习惯一个项目新建一个project,方便以后查看。
工作目录设置习惯
建议:
- 不同项目设置不同的工作目录
- 设置一个默认工作目录
- 不要把工作目录设置在软件安装目录
双击PDB文件打开PyMOL,会自动切换工作目录到该PDB文件所在目录。
从软件安装处打开PyMOL,则工作目录为软件安装目录。
下载蛋白
从PDB网站上下载蛋白,如4hbk.pdb.也可以直接通过PyMOL下载蛋白,点击菜单栏中的File->Get PDB.如下图图所示,在PDBID对应的框中填入PDB的编号就可以了,不要包含后缀.pdb。
点击Download,你会发现PyMOL会自动加载该蛋白,在工作路径D\PyMOLstartedManual下面出现4hbk.cif文件,随着PDB解析的结构越来越大,PDB格式文件的局限性就暴露出来,不能超过9999个原子,因此正在逐渐用cif 格式取代pdb格式。
介绍”ASHLC”
蛋白的展现形式(show)
在2.3对象列表窗口中,我们可以看到现在有2个object名字,
- 1个是all,all 不是真实的object,它代表了所有的object
- 1个是4hbk,4hbk就是我们刚刚载入的蛋白每个object 都有对应的ASHLC操作,如下图所示:
- Action 主要包含了对object的常用操作的集合,如复制、删除object对object加氢,展示Object等:
- Show 将object 渲染成cartoon、line、stick lines sphere surface mesh dots ribbon 等模式:
- Hide 根据object的状态或者描述进行相应的掩藏;
- Label显示object中残基原子等名称或者属性-Color对Object进行着色。
下面对Show 着重讲解:show有2类操作方法:
- show as分别点击S->as->cartoon和S->as->stick,
我们可以观察到AS模式是把原有的渲染模式抹除后再重新渲染,经过上述操作后仅仅显示stick形式 - show点击S->as->cartoon 再点击S->stick;
我们可以观察到SHOW方法,是保留原有的渲染,再添加新的渲染。
我们先对4hbk object点击S->as->cartoon.然后点击S->as->stick,效果如图Fig5
我们先对4hbk object点击S->as->cartoon.然后点击S->stick.效果如图Fig6
蛋白对象的简单平移、旋转、缩放
首先将鼠标移动到2.1可视化窗口 - 平移,按住鼠标中键不放,然后上下左右移动,进行体会,蛋白会随着鼠标而移动
- 旋转,按住鼠标左键不放,然后上下左右移动鼠标,蛋白会进行旋转
- 缩放,按住鼠标右键不放,然后上下移动,蛋白会进行缩放
- 切割滚动鼠标中键,建议将蛋白渲染成surface模式,然后滚动鼠标中键当软件不能正常使用上述操作,可以点击
File->Reinitialize->Original Settings(推荐)
File->Reinitialize->Everything 注意的是该操作会删除当前所有的Object.
Action
第一部分:常用显示操作
点击A->preset->simple显示蛋白的简单形式
点击A->preset->ball and stick 显示球棍模型
点击A->preset->b-factor putty 基于bfactor数值显示蛋白的柔性
点击A->preset->publication 高质量出版标准
第二部分对象的操作
删除水分子A->remove waters
该操作属于红色警告操作,不可逆操作。删除水分子后,无法通过Ctrl-Z进行撤销。
增加删除氢原子
在line和stick模式下面可以看到H原子,cartoon模式下面看不到氢原子,因此在line或者stick模式下,执行下述操作。
A->Hydrogens->remove 删除所有氢原子
A->Hydrogens->add 增加所有氢原子
A->Hydrogens->remove non polar 删除所有非极性氢原子,我们可以看到C上的氢原子全部被删除
A->Hydrogens->remove再次删除所有氢原子
A->Hydrogens->add polar 增加极性氢原子
第三部分对象的复制剪切删除重命名
复制A->Copy to object->new
我们会得到一个名为new的object.为了和4hbk object进行区分。
对4hbk 的obect 点击C->cyan 和S->AS->cartoon
对obj01点击C->green和S->as->stick
重命名obj01->4hbk_02对obj01
点击A->rename object
删除A->delete object
如果要删除当前所有的object的话,可以在1.3命令输入窗口输入delete ll。
剪切适合选中的对象A->Extract
我们可以通过剪切的操作把配体和蛋白分开,首先选中配体,然后点击A->extract object 就可以了,原来结构中的配体就跑到obj01中了,这样就分开了蛋白和配体。下面会有具体的例子来说明。
第四部分Action->generate操作
- 显示蛋白的静电势图Action->generate->vacuum_electrostatics->protein contact potential 就可以了。
蛋白对象的Hide操作
查看该结构的序列(Sequence)
蛋白对象的着色操作
对象的Label操作
添加label
选择需要Label的对象|然后点击对象上的L按钮,根据需要.可以标记残基的名字,原子的名字.范德华半径、元素的名字等。
- L->residue在a碳原子上标记其残基名字和编号(常用)
- L->residue name 在所有原子上标记残基名字(不常用)
- L->clear 删除该对象上所有的Label
- L->element symbol显示对象上所有原子的元素名字
- L->vdw radius产看原子的范德华半径
移动标签label
设置label的样式
调整配体的位置
pymol中包含配体和蛋白,那我们如何调整配体和蛋白的相对位置。
测量距离
点击菜单栏中的Wizard->Measurement 就可以进行距离测量。然后分别选择两个2个原子就可以显示这2个原子的距离。这里蓝色表示N端,红色表示O端。
比如我们想测定TYR48中侧链上的O原子到Lys77侧链N原子的距离。如下操作即可:
- 点击Wizard->Measurement;打开了Measurement 面板,位于object list面板下方;
- 选好残基Y48和K77,鼠标点击TYR48侧链上的O原子和LYS77侧链上面的N原子;这里为了方便,我把cartoon部分和主链部分给隐掉
- 如要测定其他2个原子的距离,鼠标继续点击选择2个原子就可以了;
- 测定完成后,点击Measurement 面板中的done就可以了。
突变氨基酸残基
其他技巧
设置透明度
鼠标操作只能基于显示模式进行设置,如:cartoon surface sphere stick等
- 将cartoon设置成透明的,透明度50%;点击莱单栏中的Setting->Transparency->cartoon->50%;
- 将Surface 设置成透明的,透明度50%;点击菜单栏中的Setting->Transparency->surface->50%;
- 将Stick设置成透明的,透明度50%;点击菜单栏中的Setting->Transparency->stick->50%;
- 将sphere 设置成透明的,透明度50%;点击菜单栏中的Setting->Transparency->sphere->50%;除了可以设置透明度外,还可以设置透明的方式,有如下几种透明模式:
- Uni-layer;点击菜单栏中的Setting->Transparency->Uni-layer
- Multi-Layer;点击菜单栏中的Setting->Transparency->Multi-Layer
- Multi-Layer(real time oit):点击菜单栏中的Setting->Transparency->Multi-layer(real time oit)
- Fast-ugly;点击菜单栏中的Setting->Transparency->Fast-ugly在Label操作中,我们将整个蛋白展示为cartoon,并将HIS等3个残基展示为stick模式;这时候我们设置cartoon为透明50%,用4种不同的透明模式渲染,效果如下:
雾化(fogging)处理
设置不同的光照模式
选择模式
根据不同的需求,切换到不同的选择模式,快速选择自己想要的原子;
- Residues残基选择模式
- Atoms原子选择模式
- Molecules分子选择模式
- chain 链选择模式
- Objects 模式
- C-alphasa碳原子模式
- Segments片段模式
我们先将蛋白Show->as wire;然后把MET-1第一个氨基酸残基show stick:如图所示,金色的表示S原子。
从上图我们也可以看到,不同模式的区别。这里我简单解释下:
保存或者导出结果
PyMOL和PhotoShop 有点类似,PyMOL中的Object类似于PhotoShop中的图层。
- PyMOL可以将会话保存为pse文件,File->Save Session;pse文件类似于PS中的psd文件,方便修改调整。
- PyMOL可以将object 导出为结构文件,File->export molecule.然后从selection的下拉框中选择需要导出的object.all代表所有的object;enable代表的可见的object;
- 保存图片,File->export image as->png;在新版本的,可以使用右上角的Ray/Trace按钮,设置图片大小分辨率,进行保存图片。
- 保存动画,File->export movie as->mpeg;PyMOL仅仅内置了mpeg_encode编码视频方式,默认只能保存mpg格式的动画。可自行下载ffmpeg.从而可以保存为多种格式,如gif.mov.mpg等
设置pse的版本号,建议版本为0.99,这样PyMOL和PyMOL2都可以打开pse文件
参考
https://wenku.baidu.com/view/628f6d6ccc84b9d528ea81c758f5f61fb73628ee.html
免责声明:本站所有文章内容,图片,视频等均是来源于用户投稿和互联网及文摘转载整编而成,不代表本站观点,不承担相关法律责任。其著作权各归其原作者或其出版社所有。如发现本站有涉嫌抄袭侵权/违法违规的内容,侵犯到您的权益,请在线联系站长,一经查实,本站将立刻删除。 本文来自网络,若有侵权,请联系删除,如若转载,请注明出处:https://haidsoft.com/129426.html