大家好,欢迎来到IT知识分享网。
火山图(Volcano plots)是散点图的一种,根据变化幅度(FC,Fold Change)和变化幅度的显著性(P value)进行绘制,其中标准化后的FC值作为横坐标,P值作为纵坐标,可直观的反应高变的数据点,常用于基因组学分析(转录组学、代谢组学等)。
一、基本概念
1、什么是FC值,为何要取以2为底的对数?
FC值全拼Fold Change value,直译为差异的倍数,在生物信息学的分析中主要用于表示基因间表达量的倍数关系。如gene1表达量为10,而gene2表达量为100,则gene2对gene1的FC即为10,由此可知FC值域为(0,+∞),如Fig. 1所示,以FC = 1为分界,绿色箭头表示表达量下调(Down-regulated),红色箭头表示表达量上调(Up-regulated),可见上调区间明显大于下调区间,当FC取浮点数时,特别是极小数时较难直观展示。反之,当倍数关系取值极大的时候还会面临上下标出界的问题。
Fig. 1 基因上下调一维展示
当取以2为底,FC的对数时,便可以解决上述问题。log[2]FC > 0时上调,log[2]FC < 0时下调。
2、为何要引入P值
P value(P值)在统计学中用于表示统计学关系的显著性,通常通过各种统计学检验获得,值域为(0,1),经校准后的P值即为Padj(P-adjected),P值和Padj值二者取其一即可。类似于方差分析,在提取有价值的基因数据点时,仅通过比较FC大小是片面且无统计学意义的,还需获得与FC值关联的P值(Padj)才可确定其关系是否显著性。同理,P值也存在类似于FC值的问题,需标准化处理,即取-log[10]P或-log[10]Padj。
通过log[2]FC值和-log[10]P的约束,即可将研究涉及的数据点放到同一个二维空间,其中离群的数据点就是我们需要关注的数据点。
二、示例
1、加载相关的包。
#绘图的包 library(ggplot2) #标记的包 library(ggrepel) ggrepel包是ggplot2的补充包,在此主要用于标记目标数据点。
2、数据准备
加载数据,此数据为python(v 3.9)生成的虚拟数据集,代码符后。在数据框中添加sig向量,用于标记上下调基因和不显著的基因,用于后续给数据点着色。
#读取数据,数据使用python随机生成,代码附后 vp_data <- read.csv("volcano_plot_data.csv",header=T) #标记上下调的基因,并添加sig向量 #上调的基因 vp_data$sig[vp_data$Log2FC > 2 & vp_data$X_log10Padj > 1.5] <- "up" #下调的基因 vp_data$sig[vp_data$Log2FC < -2 & vp_data$X_log10Padj > 1.5] <- "down" #变化不显著的基因 vp_data$sig[vp_data$X_log10Padj <= 1.5 | (vp_data$Log2FC <= 2 & vp_data$Log2FC >= -2)] <- "insig" 添加label向量,用于存放需关注的数据点的标签信息。
#为有价值的基因数据点添加lab #设置筛选条件log[2]FC = 2或-2, -log[10]Padj = 1.5 invLog10P_limit = 1.5 Log2FC_limit = 2 #abs(x)为取x绝对值 vp_data$label=ifelse(vp_data$X_log10Padj > invLog10P_limit & abs(vp_data$Log2FC) > Log2FC_limit, as.character(vp_data$Gene.name), '') 3、绘图部分
定义坐标轴。
vol_plot <- ggplot(vp_data,aes(Log2FC, X_log10Padj)) 添加数据点,根据sig列调色。
vol_plot = vol_plot + geom_point(aes(color = sig)) 自定义点的颜色。
vol_plot = vol_plot + scale_color_manual(values = c("green","grey","orange")) 添加title和坐标轴名称。
vol_plot = vol_plot + labs(title="volcanoplot1", x="log2(FC)", y="-log10(Padj)") 添加虚线标记区间。
#添加标记-log[10]Padj区间的虚线 vol_plot = vol_plot + geom_hline(yintercept = invLog10P_limit, linetype=2, colour = "grey50") #添加标记log[2]FC区间的虚线 vol_plot = vol_plot + geom_vline(xintercept=c(Log2FC_limit, -Log2FC_limit), linetype=2, colour = "grey50") 为数据点添加标签,出图(Fig. 2)。
vol_plot = vol_plot + geom_text_repel(aes(x = Log2FC, y = X_log10Padj, label=label), #添加标签图层 max.overlaps = 10000, #标签布局参数,越高标签重合越少,类似于igraph的kk布局 size=3, #字体 box.padding=unit(0.5,'lines'), #标记点边框周围的填充量 point.padding=unit(0.1, 'lines'),#标记点周围的填充量 segment.color='grey50', #标记与点的连线颜色 show.legend=FALSE) #是否添加legend vol_plot 由于是随机生成的虚拟数据,并不能真实反应实际研究中的数据分布,但绘图原理是一样的。
Fig. 2 虚拟数据生成的火山图
4、使用真实数据绘图
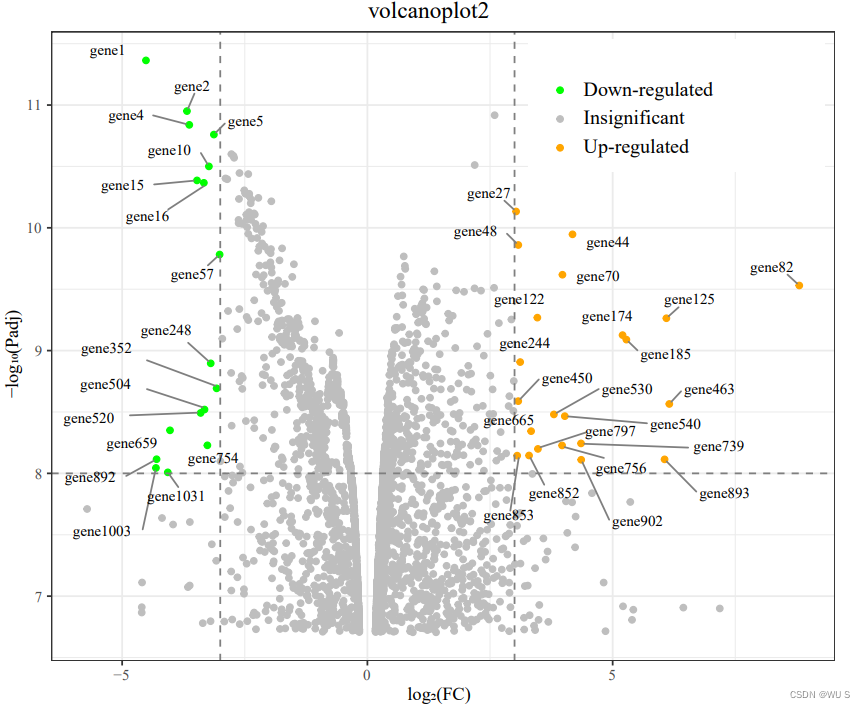
将同样的方法应用于实际数据。由于数据存在差异,将筛选条件中的-log[10]Padj改为8。代码如下。得Fig. 3。
#读取数据,数据使用python随机生成,代码附后 vp_data <- read.csv("volcano_plot_data2.csv",header=T) #更改筛选上下调基因的条件 #上调的基因 vp_data$sig[vp_data$Log2FC > 3 & vp_data$X_log10Padj > 8] <- "up" #下调的基因 vp_data$sig[vp_data$Log2FC < -3 & vp_data$X_log10Padj > 8] <- "down" #变化不显著的基因 vp_data$sig[vp_data$X_log10Padj <= 8 | (vp_data$Log2FC <= 3 & vp_data$Log2FC >= -3)] <- "insig" #添加label向量,为有价值的基因数据点添加label #更改筛选条件 invLog10P_limit = 8 Log2FC_limit = 3 vp_data$label=ifelse(vp_data$X_log10Padj > invLog10P_limit & abs(vp_data$Log2FC) > Log2FC_limit, as.character(vp_data$Gene.name), '') #绘图部分 ggplot(vp_data,aes(Log2FC, X_log10Padj)) + geom_point(aes(color = sig)) + scale_color_manual(values = c("green", "grey", "orange")) + labs(title="volcanoplot2", x="log2(FC)", y="-log10(Padj)") + geom_hline(yintercept = invLog10P_limit,linetype=2, colour = "grey50")+ geom_vline(xintercept=c(Log2FC_limit, -Log2FC_limit),linetype=2, colour = "grey50")+ geom_text_repel(aes(x = Log2FC, y = X_log10Padj, label=label), max.overlaps = 10000, size=3, box.padding=unit(0.5,'lines'), point.padding=unit(0.1, 'lines'), segment.color='grey50', show.legend=FALSE)
Fig. 3 火山图
美化后得Fig. 4, 图中灰色部分即为不显著或者变化较小的基因数据点,绿色为下调的基因,橙色为上调的基因。
Fig. 4 美化后的火山图
Supplemental
用于生成虚拟数据的python代码。
#Importing randint function to make numbers randomly from random import randint #Importing randint function to make numbers randomly from random import shuffle #A modular needed to mathematical operations import math #A modular needed to write data into .csv file import csv #demo data Gene = [] FCs = [] Log2FC = [] _Log10Padj = [] #log2FC while (len(Log2FC) < 2000): FC = randint(50, 1000)* 0.001 L2FC = math.log(FC, 2) Log2FC.append(L2FC) while (len(Log2FC) < 4000): FC = randint(50, 1000)* 0.001 L2FC = math.log(FC, 0.5) Log2FC.append(L2FC) shuffle(Log2FC) Log2FC.insert(0, 'Log2FC') #-log10Padj #Creating value spanning 0 to 1.5 while (len(_Log10Padj) < 3800): data_l10P = randint(0, 1500) * 0.001 _Log10Padj.append(data_l10P) #Creating value spanning 1.5 to 3 while (len(_Log10Padj) < 4000): data_l10P = randint(1500, 3000) * 0.001 _Log10Padj.append(data_l10P) shuffle(_Log10Padj) _Log10Padj.insert(0, '_log10Padj') #Gene name term = "gene" for time in range(1, 4001): gene_name = f"{
term}{
time}" Gene.append(gene_name) Gene.insert(0, 'Gene name') data_list = [Gene, Log2FC, _Log10Padj] #List transpose data_list = list(map(list, zip(*data_list))) #Writed list into a file.csv with open('volcano_plot_data.csv', 'w', newline='') as file: writer = csv.writer(file, delimiter=',') writer.writerows(data_list) Ending!!!
免责声明:本站所有文章内容,图片,视频等均是来源于用户投稿和互联网及文摘转载整编而成,不代表本站观点,不承担相关法律责任。其著作权各归其原作者或其出版社所有。如发现本站有涉嫌抄袭侵权/违法违规的内容,侵犯到您的权益,请在线联系站长,一经查实,本站将立刻删除。 本文来自网络,若有侵权,请联系删除,如若转载,请注明出处:https://haidsoft.com/142321.html